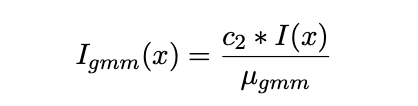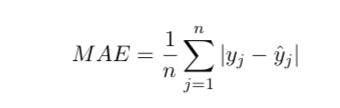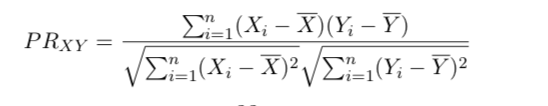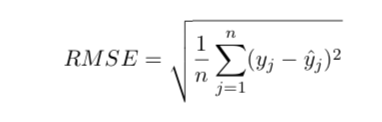今天是武汉肺炎在家里宅着的第五天,想起来想写的博客里有个坑还没填上,就是我的毕设,在之前的博客里,讲了项目梗概、图片处理方法等等,然后就没下文了。如今毕业已经4个多月了,已经完全将这件事抛之脑后,如果这个时候都不把它写上,以后一定就不会再去管这件事了。我不想成为一个做事情虎头蛇尾的人,所以想在这篇文章里梳理一下整个项目。
总的来说,我的毕设完成度还是相当高的,做literature review花了一个学期,正式毕设花了大约3个月,答辩完成后得到了80分的成绩(英国教育下给分严格,50分及格,80分优秀),最后硕士也给了distinction的评价。
因为内容太多,所以这里只是做一个概述,如果有兴趣阅读全文的话,原文链接在这里,答辩ppt在这里。代码在这里,以前写的代码有点乱,也没写注释什么的,见谅。
简单来说,项目目标就是给定一张3D的MRI脑图,预测其年龄。文章将分为以下几个部分:
- 背景知识
- 数据收集
- 图像预处理
- 传统机器学习模型
- 深度学习模型
- 结果
- 服务部署
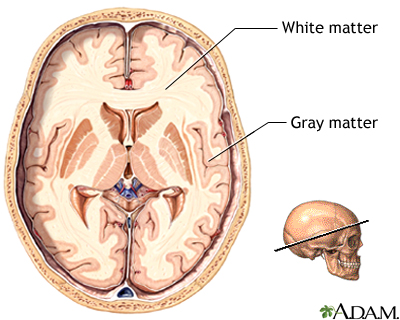
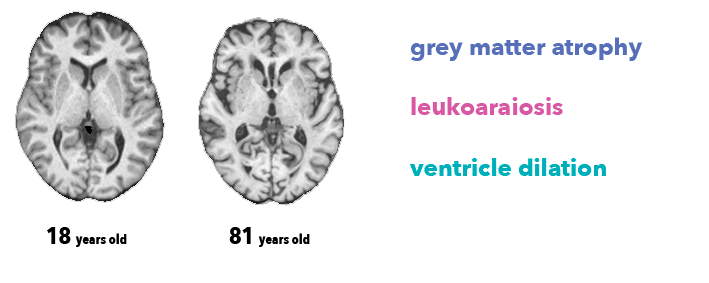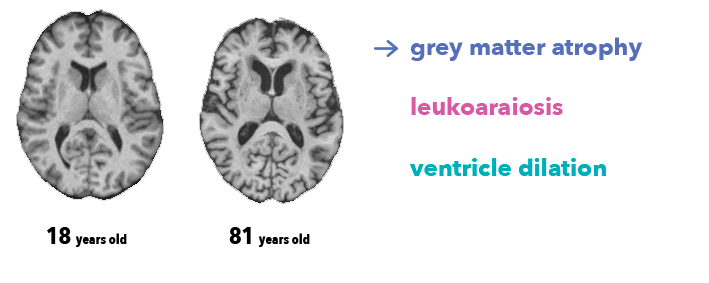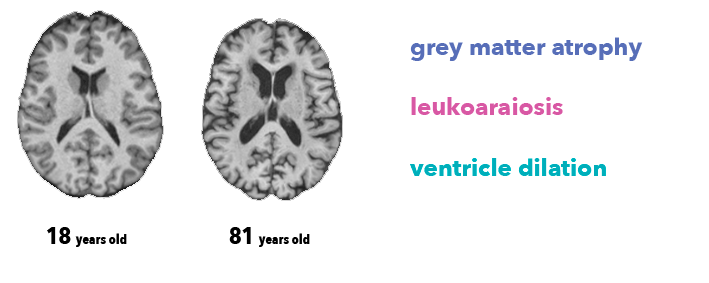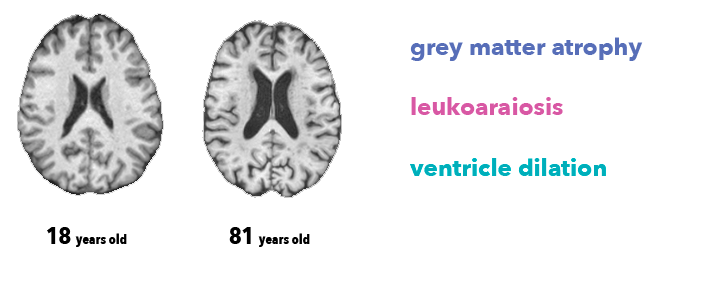
众所周知,人的大脑会随着人的年龄增长而逐渐衰老,因此人脑和年龄就会产生某种联系,脑灰质和脑白质会疏松,而脑脊液会在脑室积聚,如图所示:

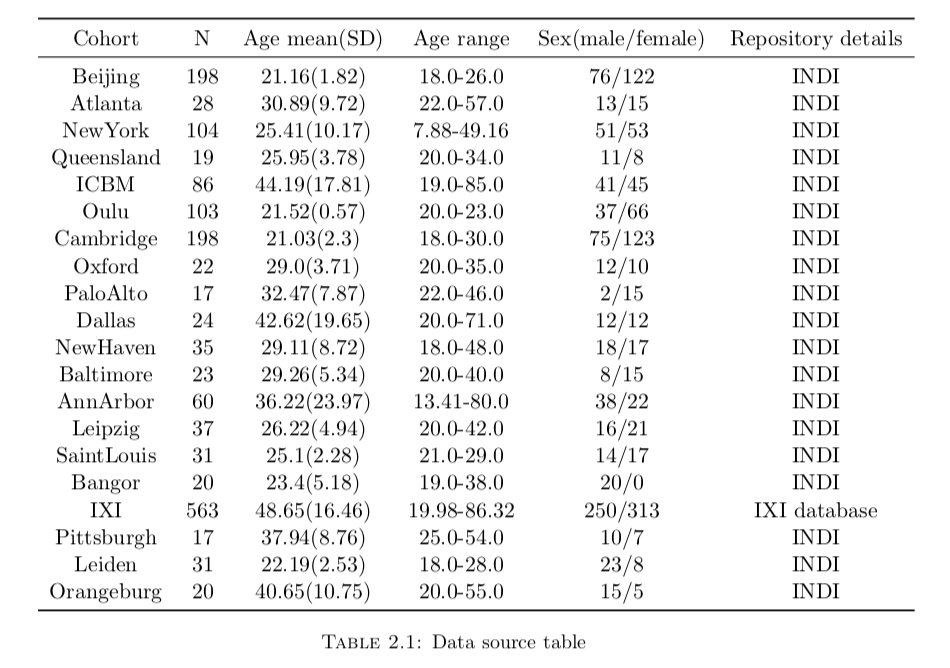
该项目共使用了1635个样本,这些数据有些是学校、医院里面的公开数据,也有一些是需要申请才可以获得的私密数据。总之我把它们都放在了 [数据源](https://storage.cloud.google.com/dissertation wzy/raw.zip),需要翻墙才可以访问,解压完以后总共大约60G左右。
另外每个数据源都提供了一张表,描述了每个图片对应的年龄,但这些数据有部分缺失或者明显错误等等,所以需要做一些数据清洗工作。
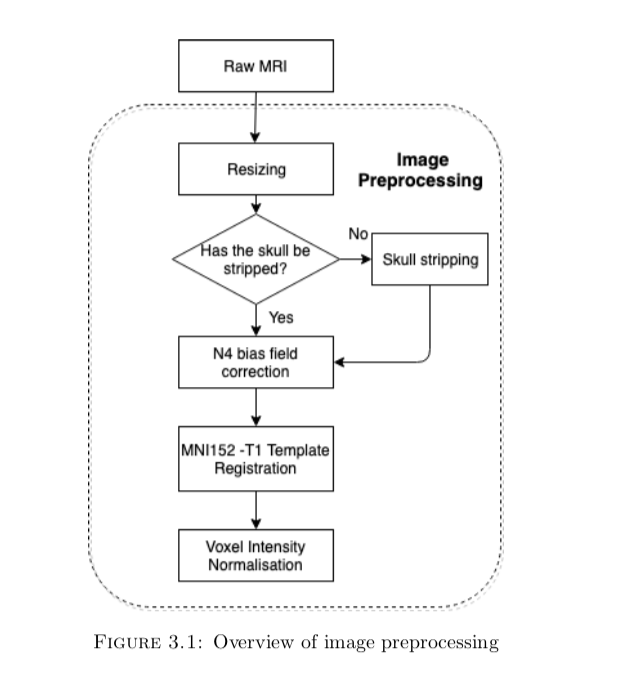
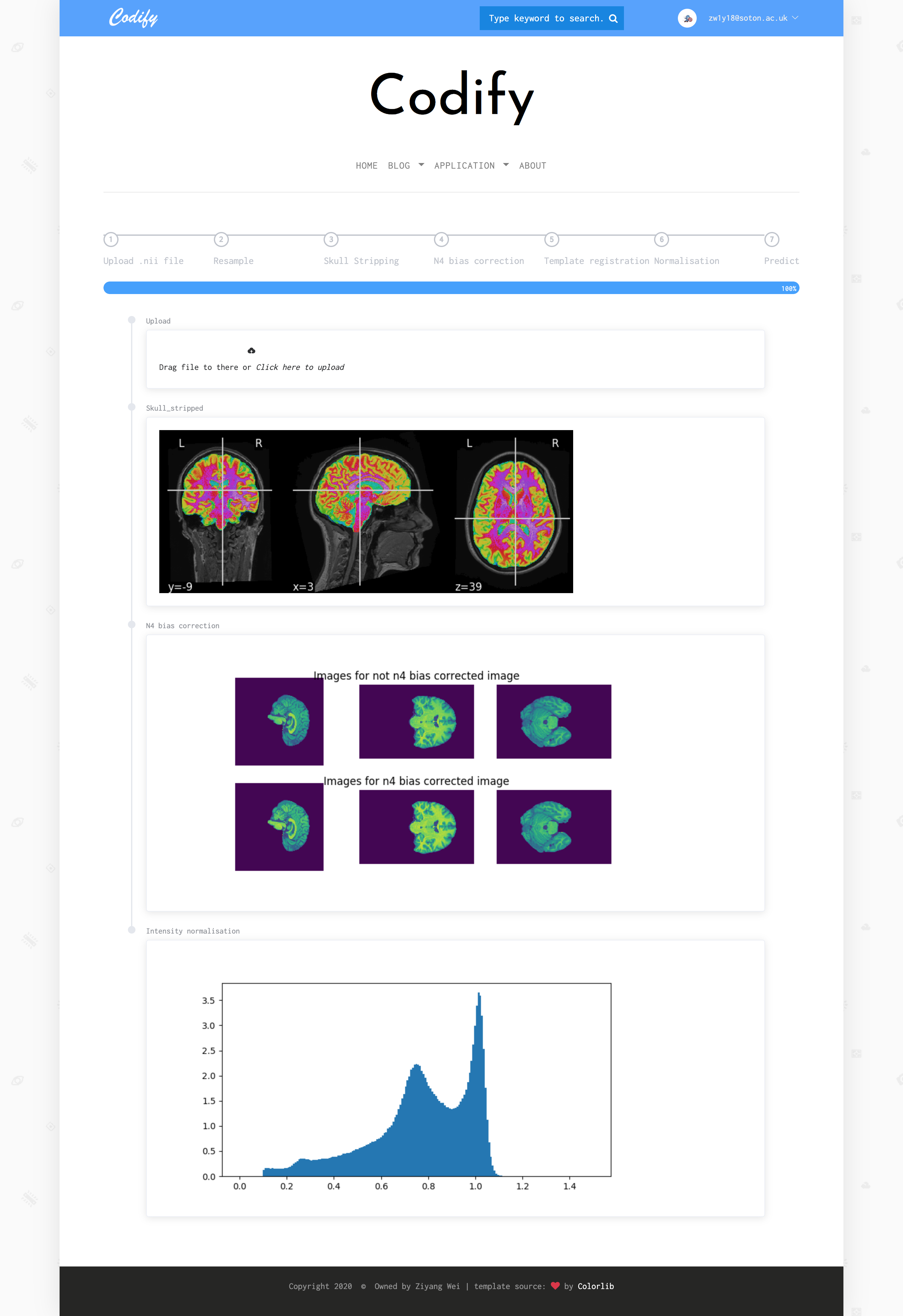
这些采集的图片由于设备、分辨率、对比度、视角、方向的不同,是不能直接用来训练模型的,因此需要做一些图片预处理(之前踩的大坑踩在了这里,没有做n4 bias field correction,导致最后的误差永远在15岁以上),预处理的流程如下,这个过程已经相当于业界的通用做法了。:
由于每个设备的参数不一样,会导致图片的分辨率产生很大的差异,简单来说,一个voxel(pixel在三维世界中的表示)所代表真实世界的长度不一致。由于每个MRI图片的meta data 都提供了分辨率这个参数,根据这个参数去进行统一,所以这个实际上很好办,这里我们就将分辨率直接归一化为111了,从而尽量使图片信息保存的比较完整。
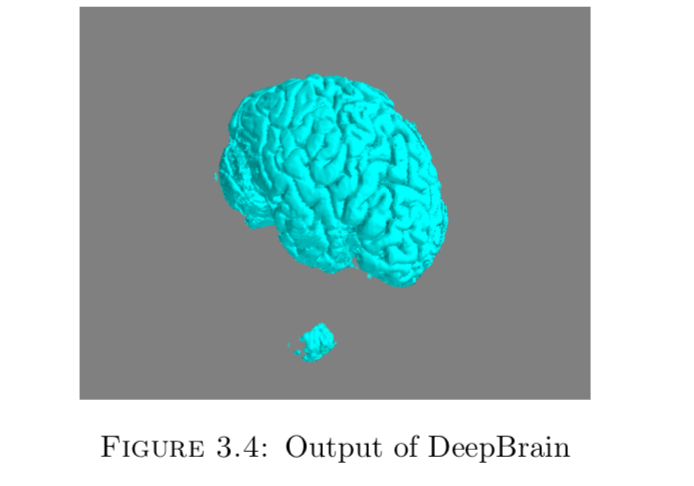
之前给的示例图片大家也发现了,这丫根本不是一个脑子,而是一整个头啊,而我们实际上需要的仅仅是脑部这一小部分,所以这步非常必要。这里我用了一个预训练好的的Unet来进行切分github地址
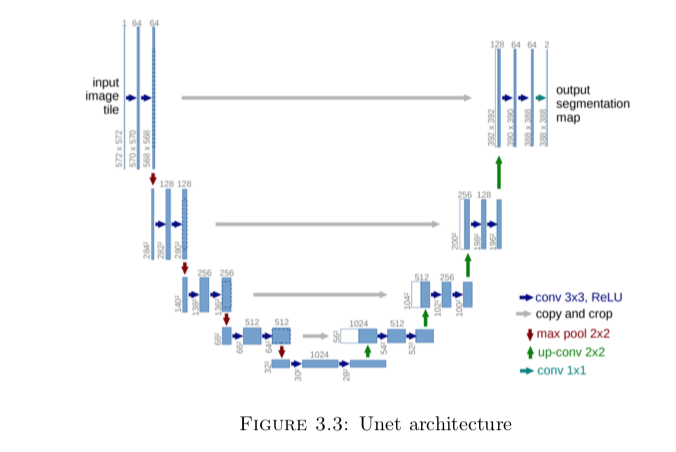
而Unet的整体结构是像这样,大致的意思就是不断的downsampling, 然后再upsampling回去,把相同层的结果相加起来,最后输出分割图像的概率。其实我觉得unet和resnet从某种程度上来说,非常像。
可以看出还是有点瑕疵的,下面有些小blob没有被分割出来。这里我们用morphonlogy形态学的方法来处理一下,已知脑部是最大的连通区域,其他所有的连通区域的体素点数量一定没有脑部多,所以直接把这些连通区域删除就行了。算法上一个深度优先遍历就可以搞定,所以最后出来的图像像这样:
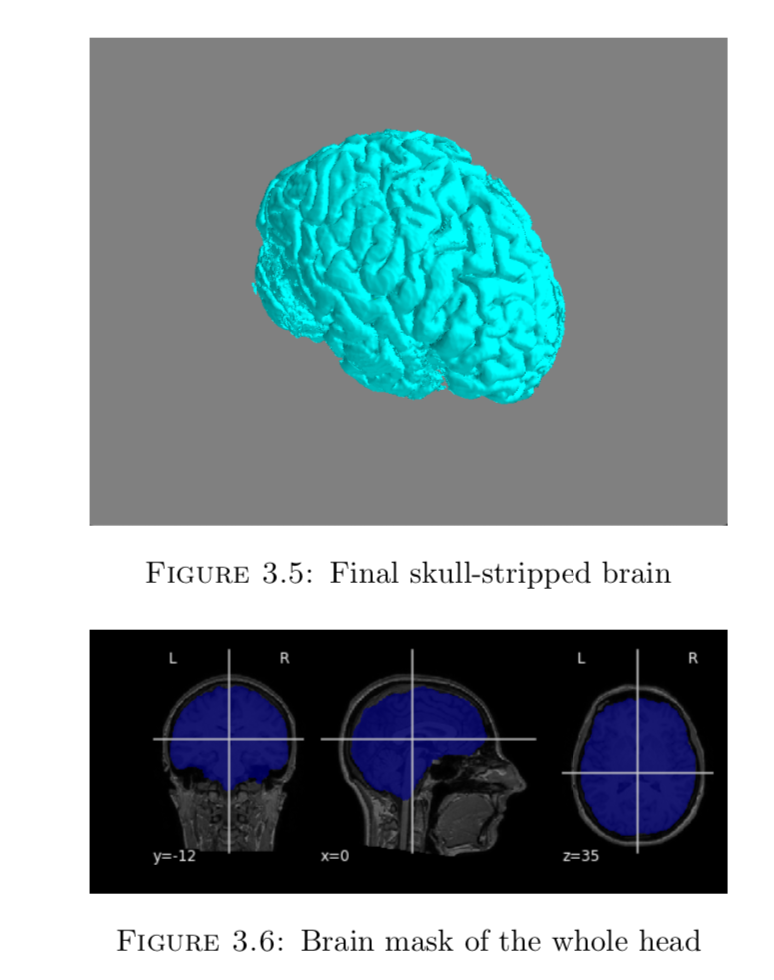
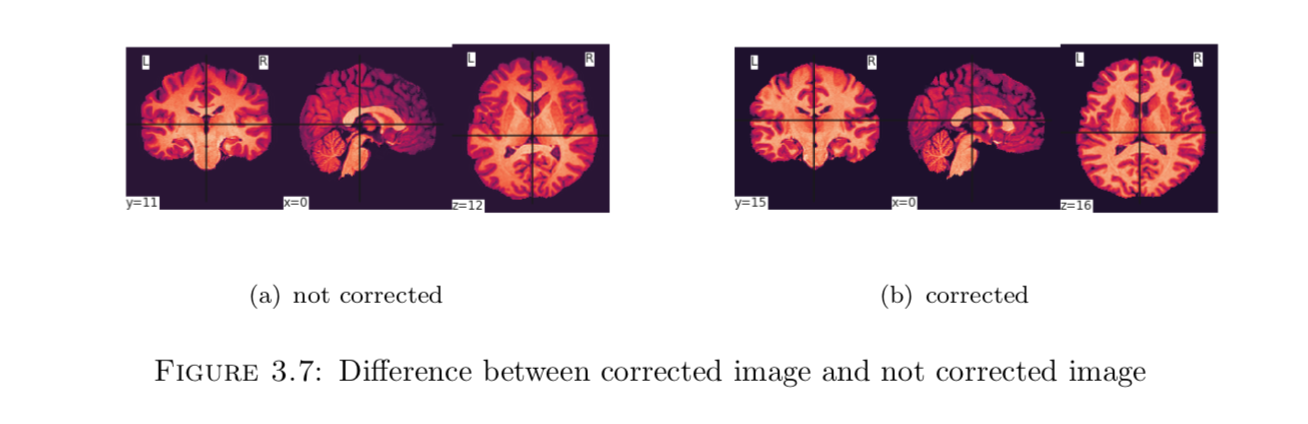
第一次踩坑就踩在这里了,因为自己缺乏对医学影像的知识,所以忽略了这个步骤。所谓偏移场信号指的就是一种低频光滑对MRI脑图造成破坏的一种信号。这个信号是由机器自身施加的,所以无可避免,可以看一下偏移场是如何破坏一张图片的:
左边是被破坏的图像,右边是修正后的图像。可以看到被破坏的图像中,脑白质部分的像素点强度不均匀,在脑灰质附近的脑白质像素点强度明显暗于其他地方的脑白质像素点强度。
而修复方法也不需要我们自己去进行更多的研究了,Tustision提出了一种基于B-spline 近似的方法去消除这种由偏移场引起的不均。SimpleITK库提供了实现方法,由于这个算法及其复杂,需要迭代很多轮,所以平均处理一个图像需要6min时间,所以1600张图片总共花了大约5天来处理。
由于每个医院提供的脑图的形状、方向都不一样,所以在喂进模型之前,还需要进行图片配准工作。2D图片有transform矩阵进行平移旋转、3D图片也是有的,但其transform matrix 会更为复杂,具体可以看论文原文,里面介绍了3D图片的所有transform矩阵。
而配准需要一个template为基准,这里使用的是MNI152 standard-space T1-weighted average structural template,最后图片将被归一化到182*218*182。详情翻原文
最后template registration可以用牛津大学提供的工具:FSL来做。
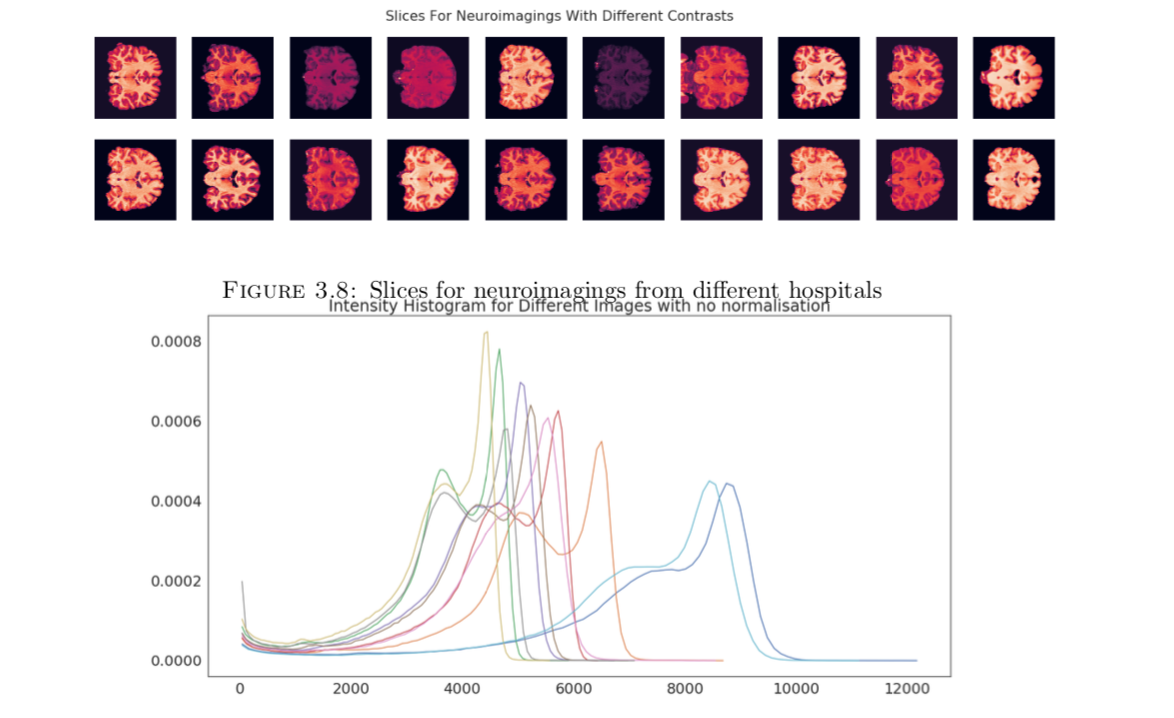
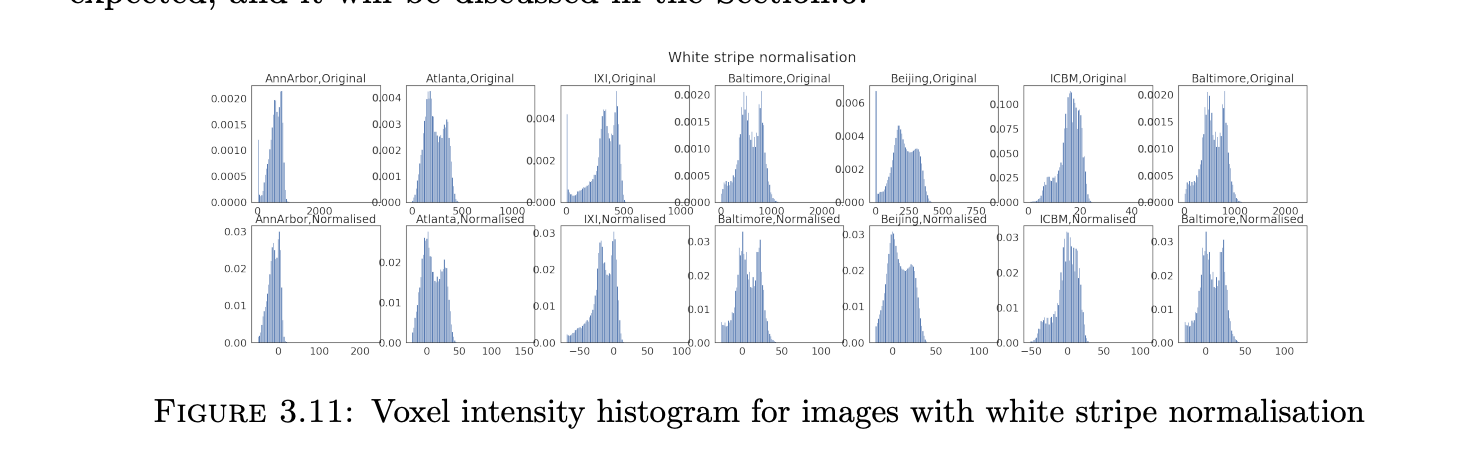
由于设备的参数不同,因此图片的强度范围也大不相同,下图展示了各来源不同图片的切片,而3.9 则是灰度直方图的对比。
归一化的方法有很多种,不同的方法也会对最后的结果产生不同的影响,详细看这篇论文:intensity normalisation。
Z-Score是其中最简单的一种方法,它把所有体素的均值和方差算出来后,用每个体素的值减去均值再除以方差,最后可以获得一个[-1,1]间的数字。公式如下:
所谓GMM 的全称就是Gaussian Mixture Model,高斯混合模型了。他做出了这样一个假设:MRI脑图的灰度直方图是由三个不同高斯分布组合而成的,而这三个高斯分布分别对应的就是脑灰质、脑白质、脑脊液。
具体归一化公式如下:
其中C2是个常数,I(X)是像素值,μ_gmm指的是均值最大的高斯分布的均值,也就是脑白质的均值。如此可以将所有脑白质的部分归一化到1.最后形成的图像如下:
White-stripe 跟GMM有点类似,它也是先去找到脑白质所对应的像素值,然后将图形像素点减去脑白质的均值,再除以脑白质的方差。但是它跟GMM不同的地方在于:1,首先它的用的模型不是高斯混合模型,而是KDE,也就是核密度估计,关于核密度估计和高斯混合模型的数学原理在这篇文章里面不展开叙述,有兴趣的可以看我代码实现。2,其次GMM只取了脑白质的峰值,是一个单一数值,而white-stripe包括了峰值附近的所有脑白质。3. 它是将脑白质部分归一化为0,不是1。
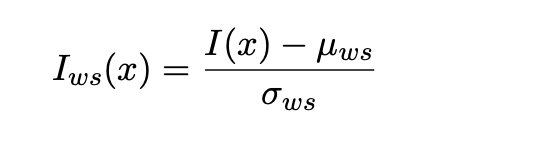
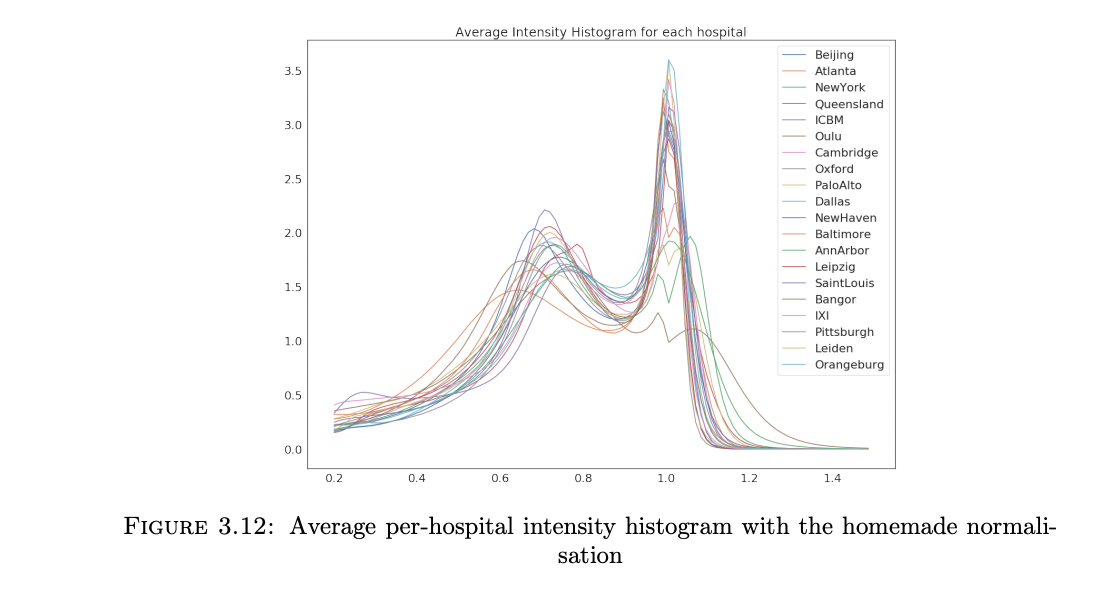
这个方法是我从一篇博客里面看到的,博主是法国的一名数据科学家,他发现了一个问题,就是以上所有的方法都只能将脑白质的部分固定到一个固定的数值,而脑灰质的数值则没有固定,所以他提出了一个方法来将这两部分分别固定在0.75附近和1。方法如下:
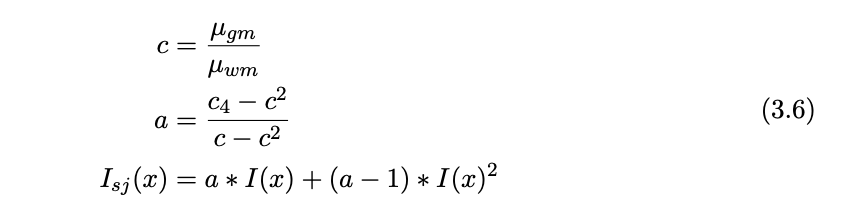
c是根据脑灰质的平均值除以脑白质的平均值得到的,c4是一个常数,这里给它0.75。最终效果如下:
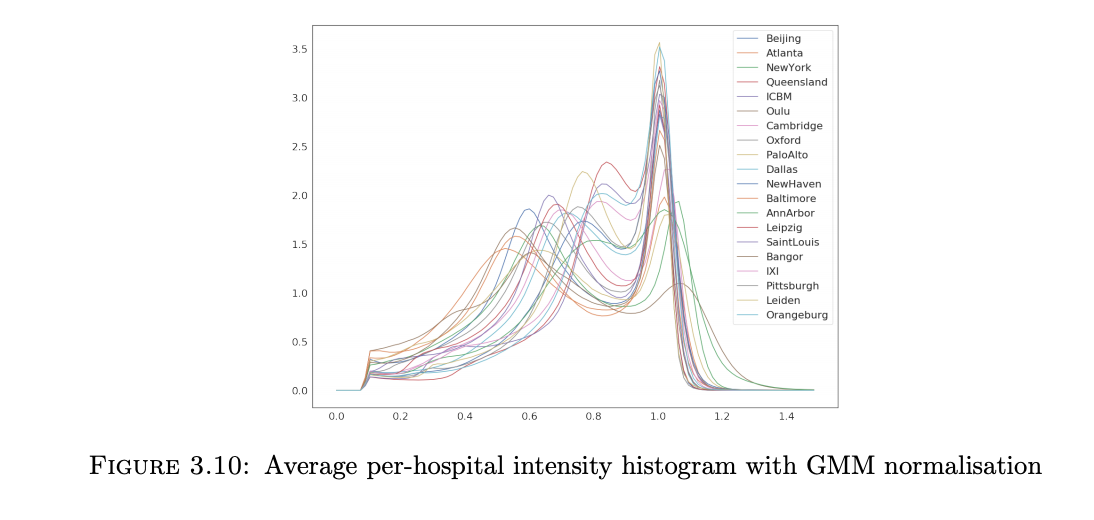
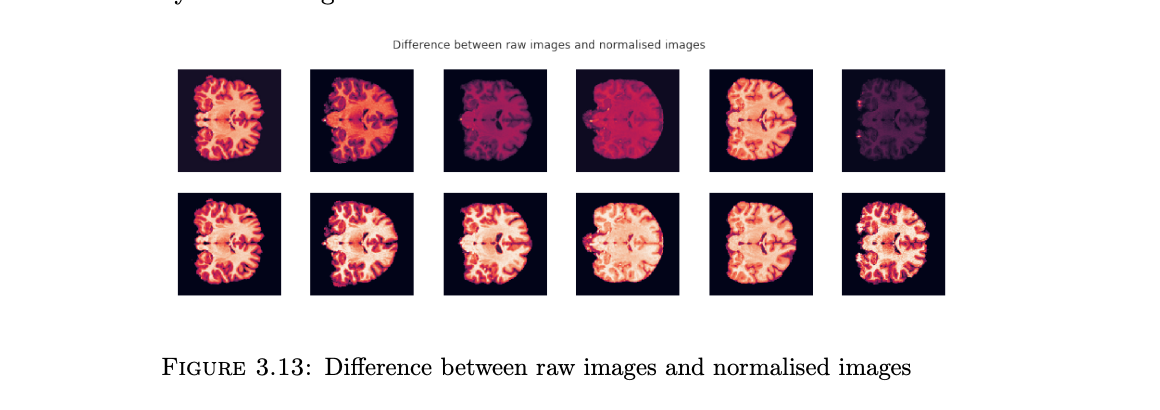
下图就是最终归一化的结果了,上面一排表示了未进行归一化的图片,下面一排表示的是用GMM归一化后的图片,可以看出效果很不错的还是。
一般来说,传统的机器学习方法首要任务就是进行特征提取,那么对于这个任务来说,如何对三维图像进行特征提取呢?
首先,我们要清楚,最终输出的年龄和什么有关,先前我们提到过,和脑灰质、脑白质、脑脊液的含量是有关的,那怎么去表示这些含量?
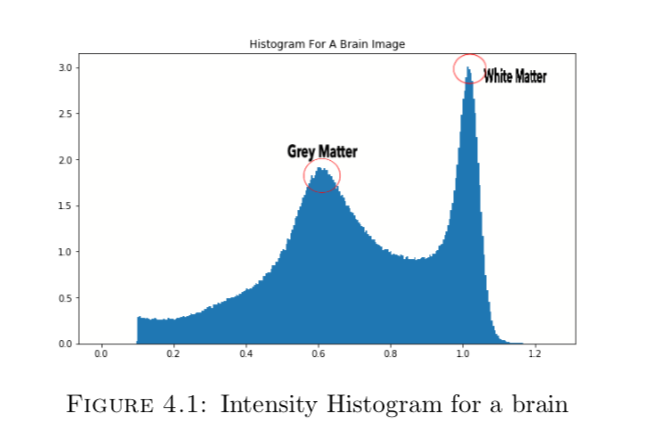
可以看到,有两个高峰,其中第一个高峰就代表的是脑灰质(因为颜色偏暗,所以数值低), 第二个高峰代表的就是脑白质了。我们可以把高峰所对应的数值那块给mask掉,来检验我们的理论是否正确。
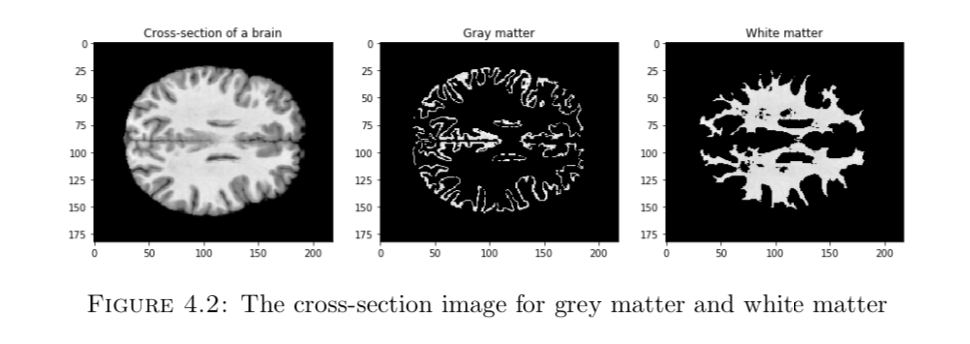
事实表名非常吻合我们的假设,所以我们可以直接把直方图分为100份,每份对应的数值就是一个特征值。
模型结果的验证方法用了比较经典的十折交叉验证法,这里衡量模型的好坏用了以下几种标准:
MAE就是平均绝对误差,非常直观,这个值越小越好:
Pearson Correlation Coefficient,是皮尔逊相关系数,代表两个变量的相关程度,这个值范围在[-1,1]之间,大于0代表正相关,小于0代表负相关,这个值越大越好。
R2也就是决定系数,用于度量因变量的变异中可由自变量解释部分所占的比例,以此来判断统计模型的解释力。公式可以自行谷歌,这里不赘述。范围在[0,1]之间,越大越好。
RMSE,均方根误差, 也是用来衡量误差的一种方法,但是这种衡量方法对大误差更为敏感,该值越小越好。
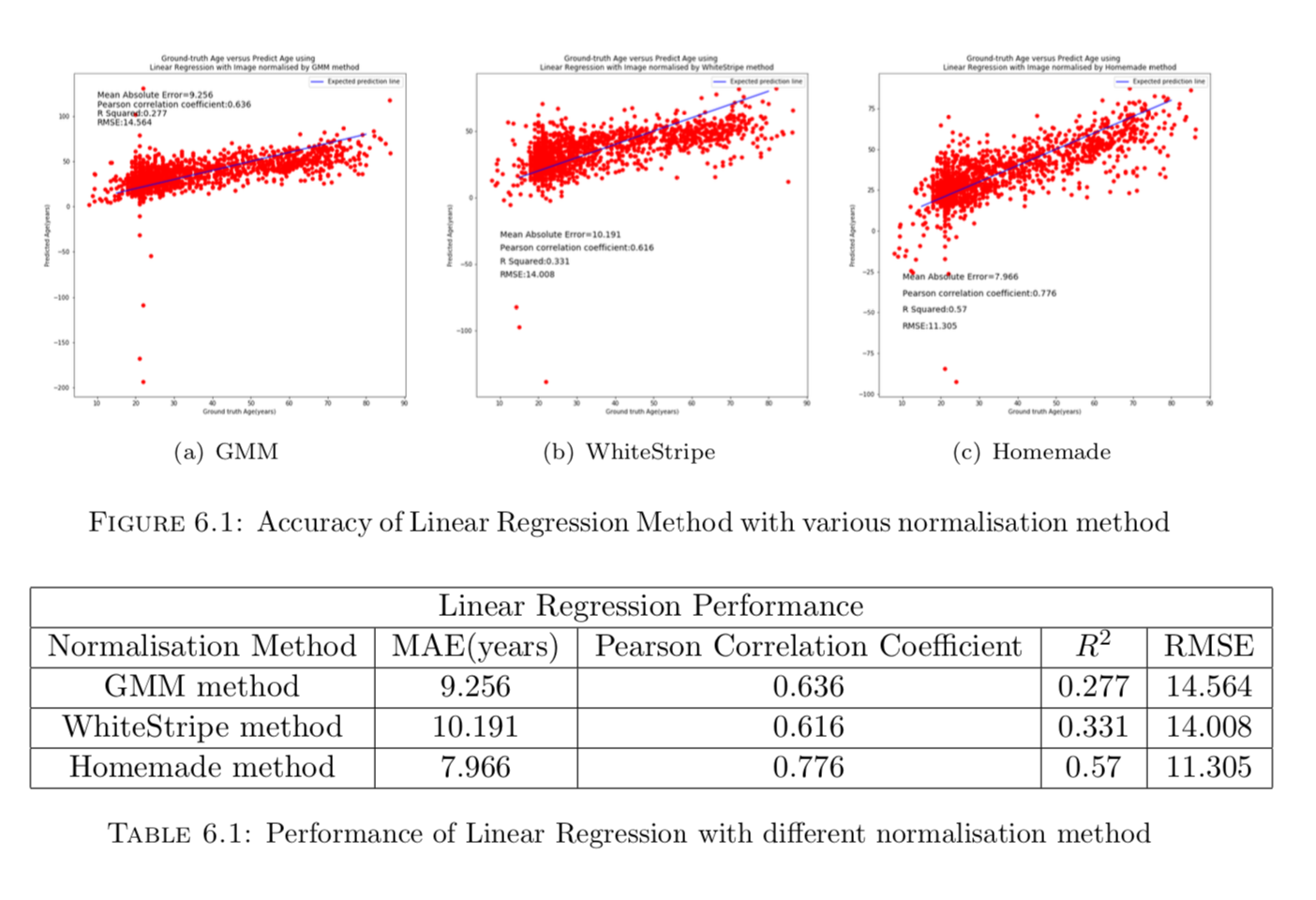
先从最简单的模型开始做起,线性回归,关于线性回归的数学理论在这里不做叙述了,可以直接翻论文原文看,简单来说线性回归是用于确定两种或两种以上变量间相互依赖的定量关系的一种统计分析方法。
先前说过,不同归一化方法会产生不同的结果,由于Z-Score没有把数值压到一个固定范围以内,所以就不做Z-Score的对比了(效果太差),其他结果如下,Homemade 其实就是Simon Jgou’s 提出的归一化方法:
可以看出线性回归并不能很好地拟合这个特征,并且预测的结果中,出现了类似例如-200这样的异常值,原因在于灰度直方图和其真实的年龄并没有出现很好的线性关系。
其实这之前,我还用了高斯过程回归,可以在论文里面看到,但是我后来仔细瞅了瞅,感觉这个场景应该不适合用高斯过程回归来做,或者说,别的相关论文里面用的高斯过程好像用的特征跟我的不一样,所以高斯过程的效果其实也不太好,所以这里我就不提高斯过程了,直接跳到随机森林了。
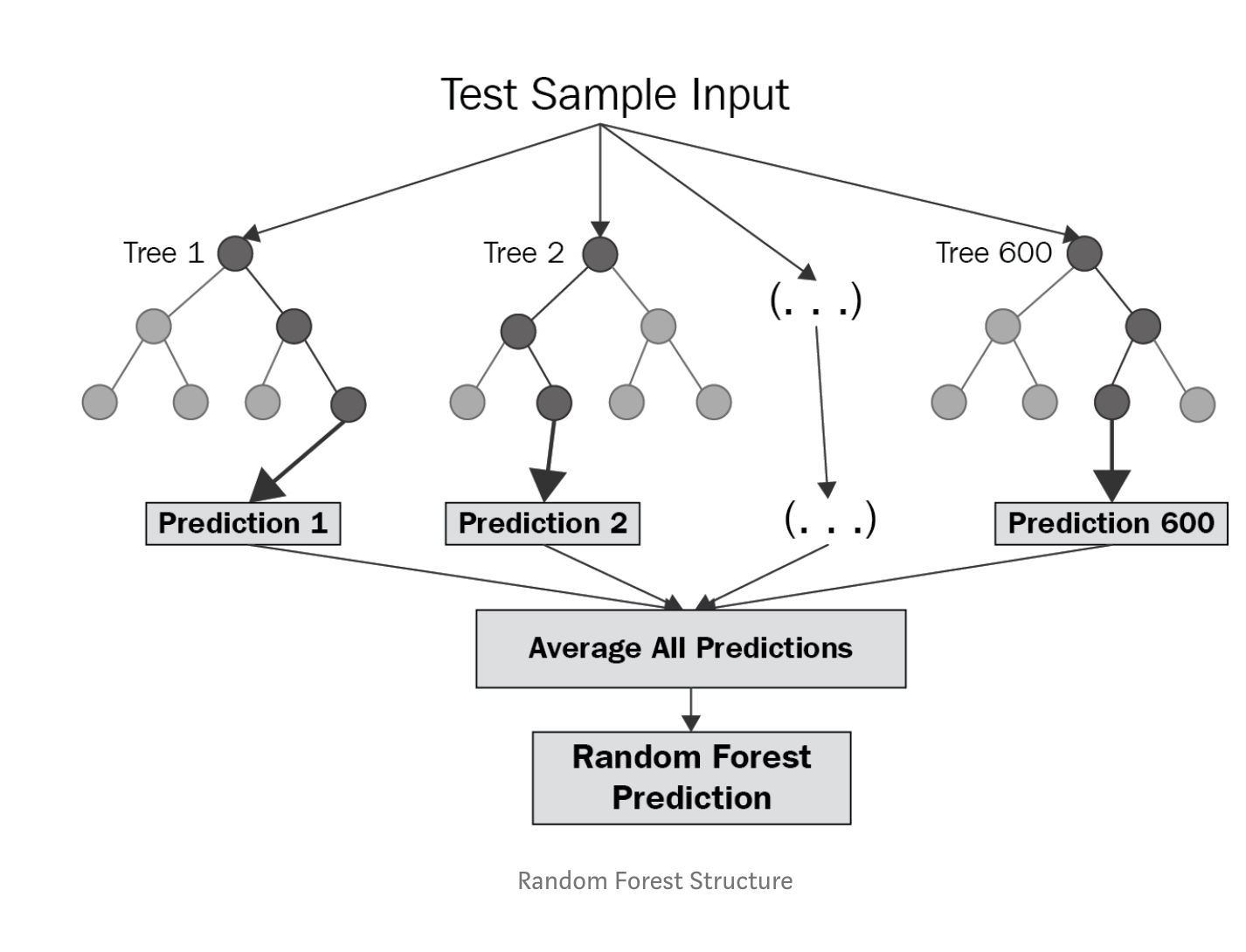
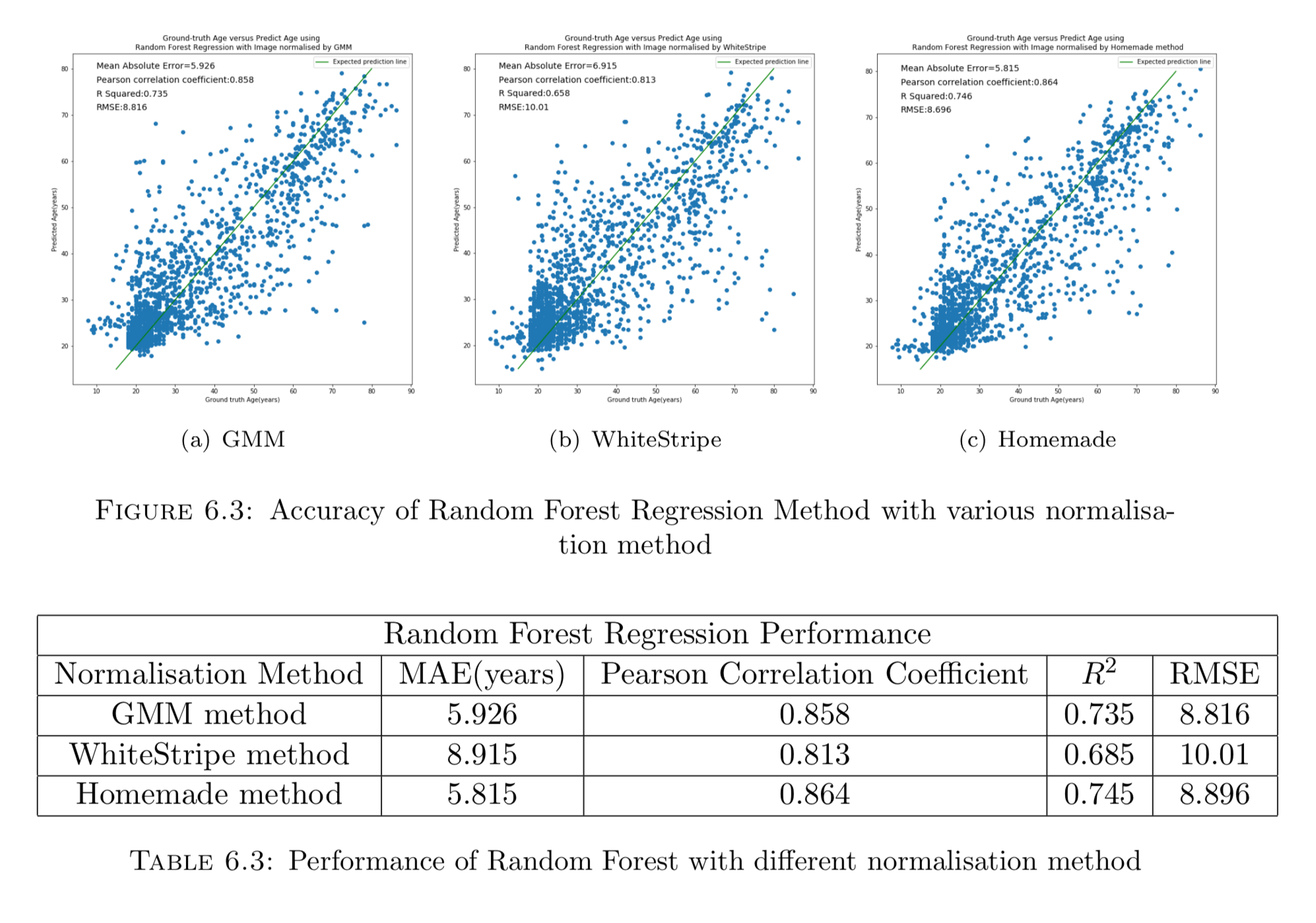
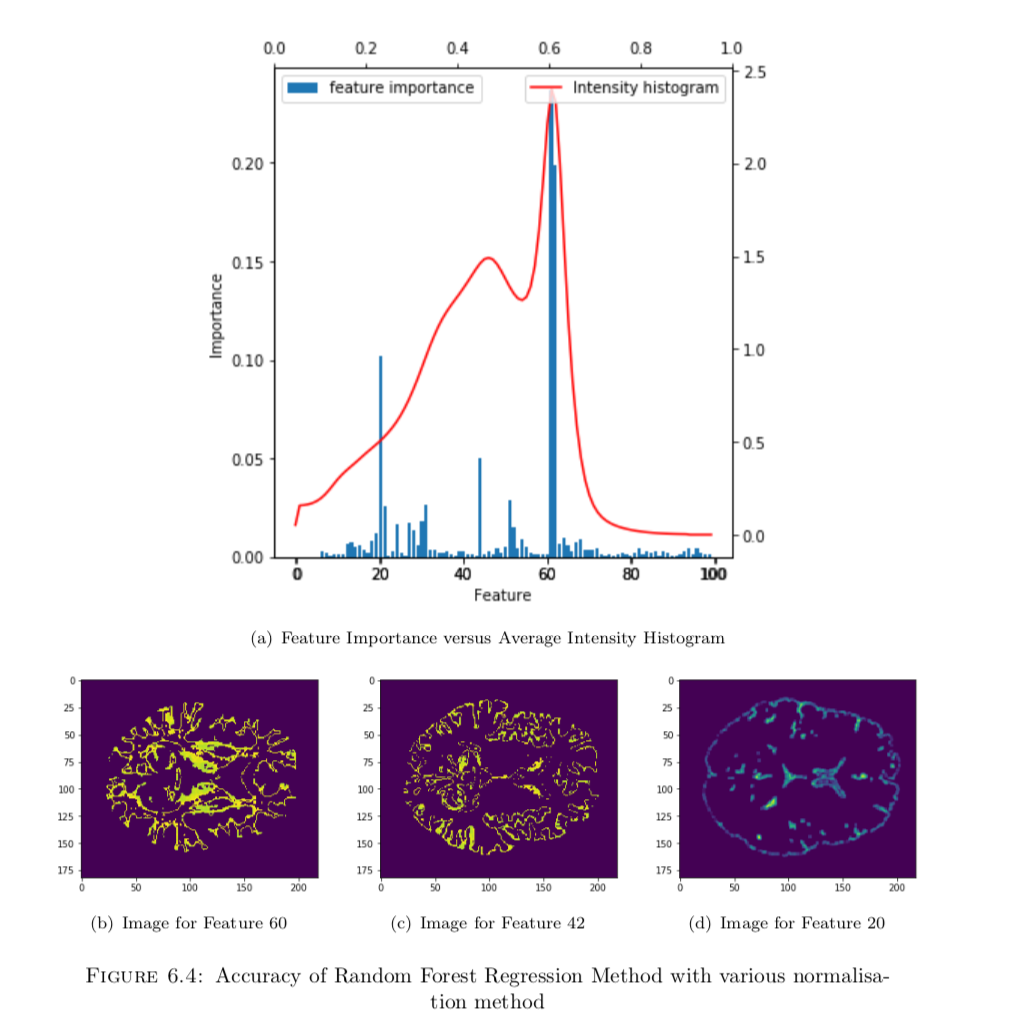
实际上随机森林是我最后写完论文快定稿的时候,别人推荐我用这种方法来试的,本来传统机器学习的方法是被深度学习直接碾压的,但用了随机森林以后,竟然发现效果还不错,误差比深度学习仅仅高了1岁,并且这种方法能够揭示一些有趣的东西。
提到随机森林,实际上就是多个决策树ensembling的结果,每个决策树都只取部分样本的部分特征,随机森林的随机就体现在这儿,因为每一个决策树都获取了一部分信息,所以将所有决策树预测出来的结果求个average,就是最终结果了。它实际上就是用多个高variance模型集成后的结果来降低总体的variance。
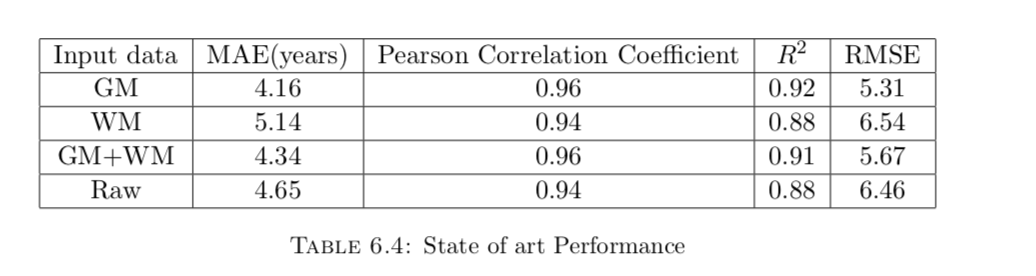
目前的state-of-art是用深度学习训练出来的,最终误差为4.16年,而我们的随机森林可以达到5.815年,实际上已经是一个很不错的结果了。但RMSE仍然有些大,对高年龄的预测结果并不是很好。
另外由于随机森林和决策树一样,可以揭示出特征的重要性,所以我们可以这样玩一玩,求出所有图片的intensity-histogram的平均值,然后和特征重要性的柱状图来进行比对,看一下有没有什么发现:
深度学习在CV领域已经几乎一统江山了,很多state-of-art都是用NN刷出来的,而该项目最后的最好结果也是用深度学习做出来的。
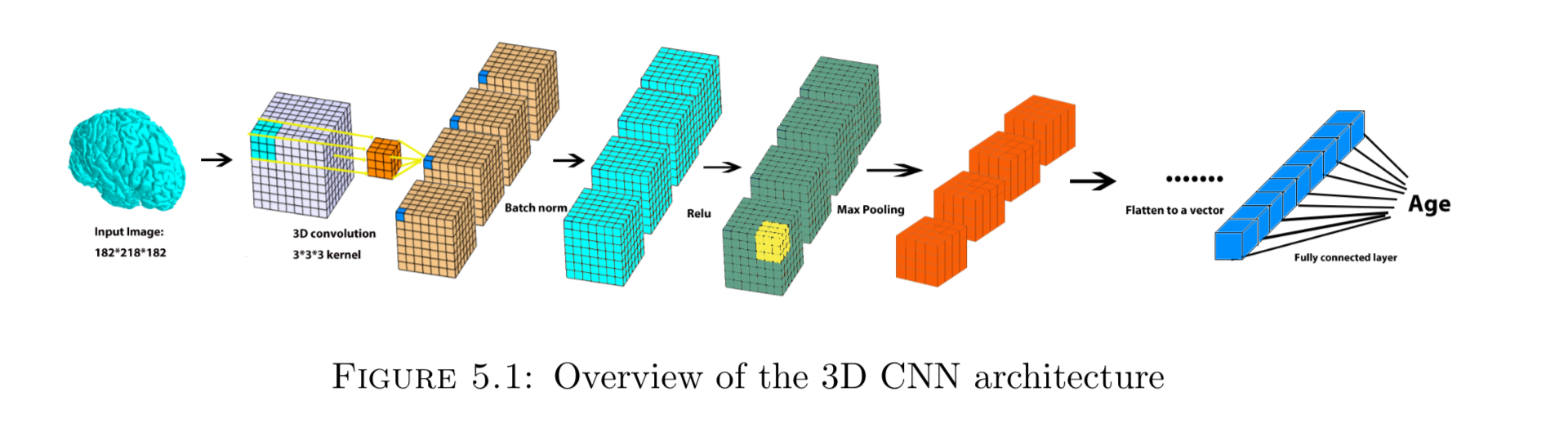
和普通的CNN不同,由于MRI脑图是三维图像,所以需要对传统神经网络做一点改造,大致结构如下:
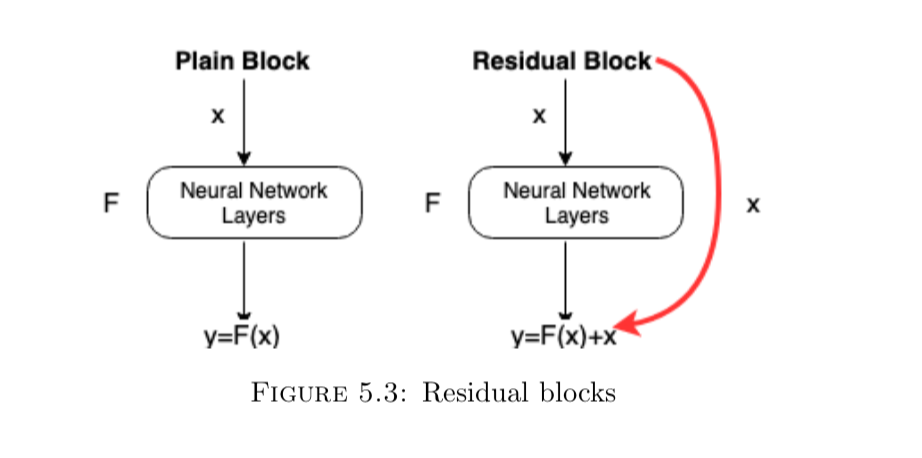
这里就不再说明,conv层、batch-norm、pooling层、relu等的作用了,不懂的翻翻其他博客补一下基础。由于神经网络会随着深度的增加而出现梯度消失的问题(一层一层地对F(x)求导做连乘,最后乘出来的数字将会无限接近于0),所以出现了resnet,通过加一层shortcut,使得求导出来的值恒定大于1,从而很巧妙的解决了这个问题,下图是resnet的residual block示意图:
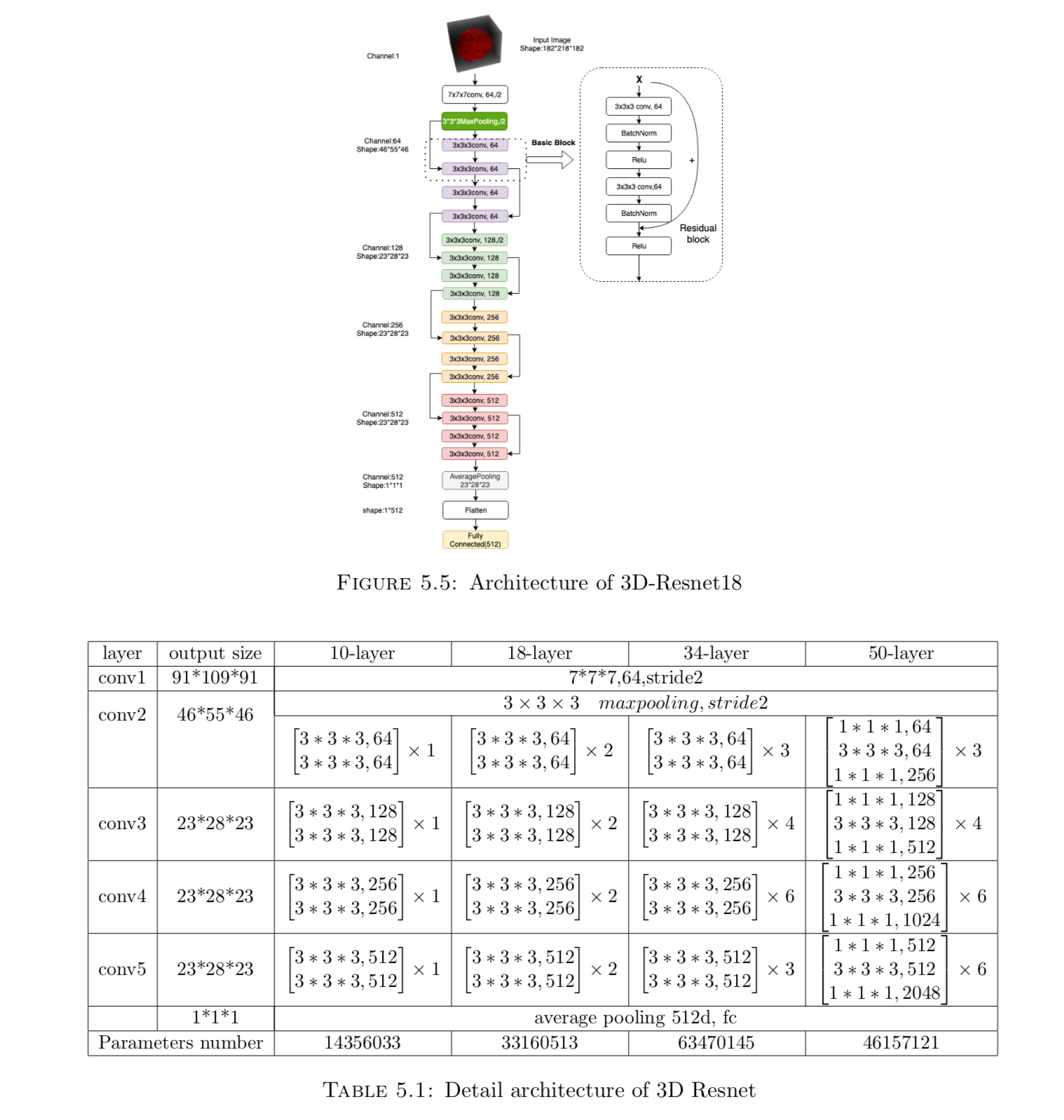
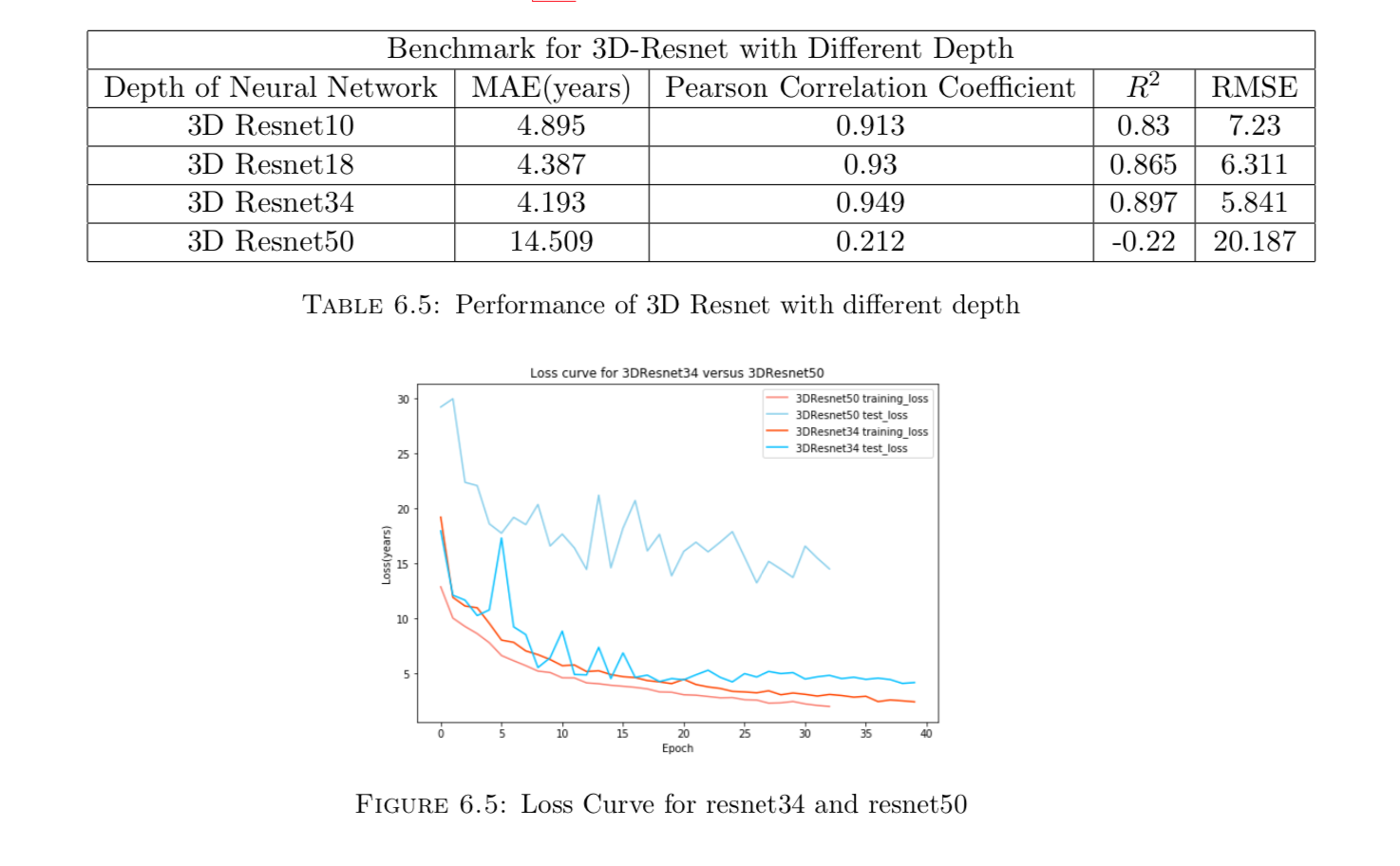
由于是回归预测问题,所以我们使用MSE(Mean squared error)作为损失函数,Adam作为optimizer,参考了Sihong Chen, Kai Ma, and Yefeng Zheng. Med3d: Transfer learning for 3d medical image analysis. arXiv preprint arXiv:1904.00625, 2019论文中提出的结构,结合该项目需求,我搭建了resnet10, resnet18, resnet34, resnet50最终的网络结构如下:
训练了大约四五天吧,每个结构跑100个epoch,最终结果如下:
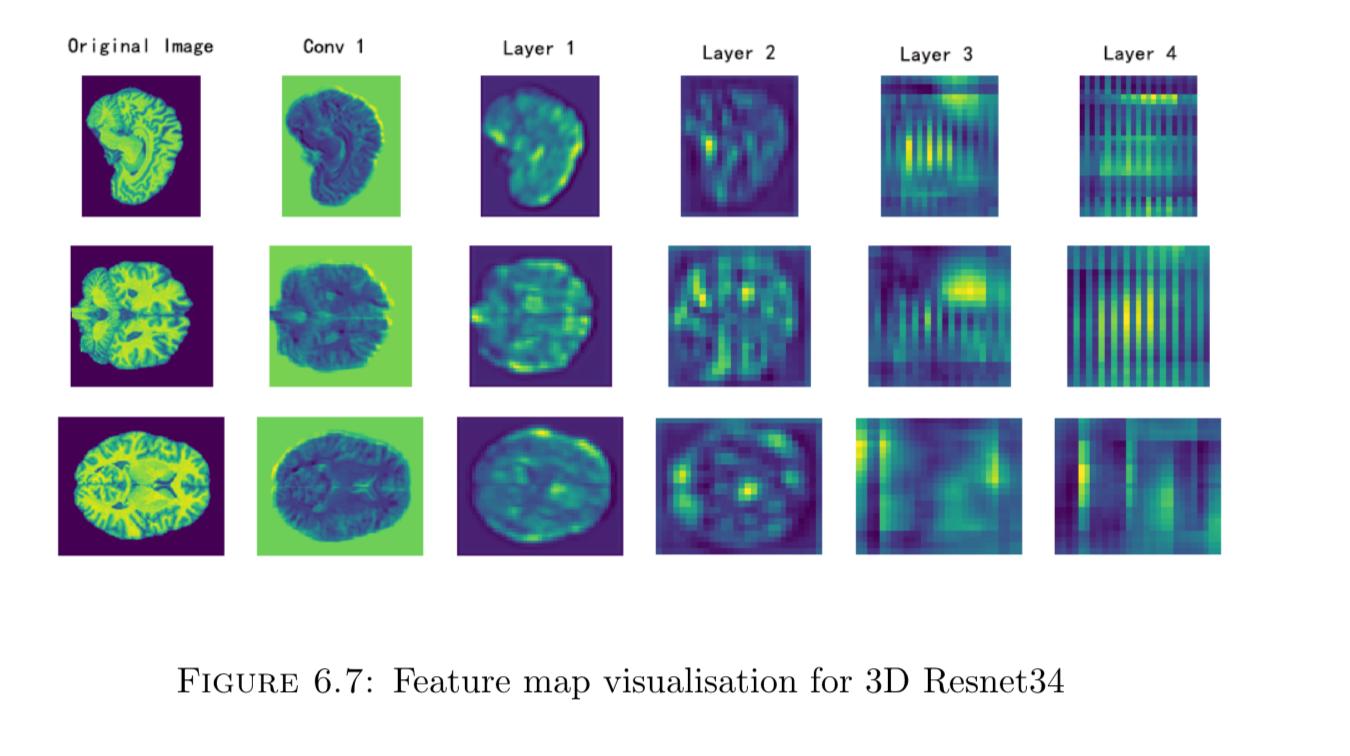
最后把resnet34每层的卷积结果抽出来做feature map visualisation,结果如下:
好像没得到什么有用的信息,但实际上有很多种方法可做feature map visualisation,很遗憾我没有去做更深一步地探索。所以特征可视化就到这里吧。
最后我把项目做了一个不成熟的部署,地址 https://www.wzy-codify.com/age-predictor, 用户可以提交.nii MRI图像文件,然后它会自动去做图像处理的pipeline 并实时反馈给用户,最后给出一个年龄预测值。
还有就是我做这个项目时,也不知道工业界里做inference不能直接把数据丢pytorch里面跑,后来加入一家NLP独角兽公司,才知道用tensorRT会更好一点。所以部署的这个玩意儿看起来傻傻的。
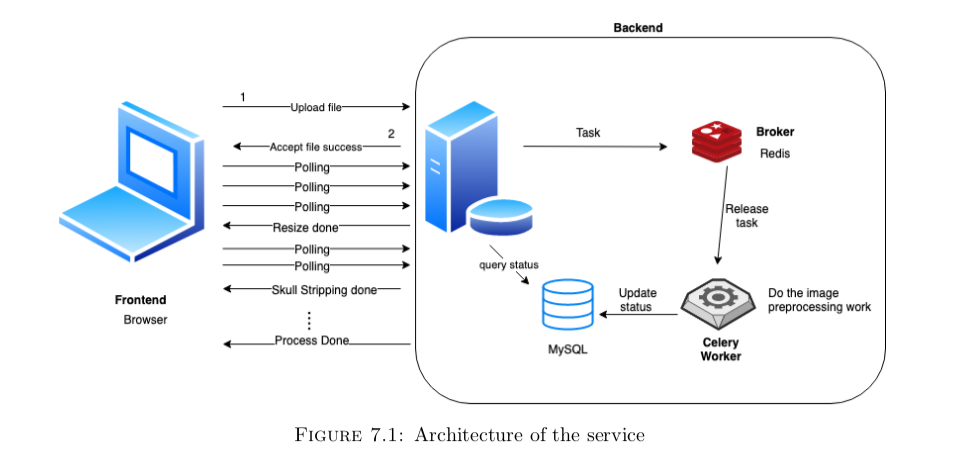
根据项目需求,这是一个异步任务,所以我后端采用了celery + redis的方式来进行处理。结构如下:
- 复现了MRI图像处理的pipeline,包括image resizing, skull stripping,n4 bias correction, template registration, 以及 intensity normalisation.
- 复现了四种不同的Normalisation方法。
- 尝试了三种不同的传统机器学习方法并比较他们的优劣。
- 通过随机森林的feature importance,揭示了与年龄最为相关的特征——脑白质含量
- 实现了一种 3D resnet 来进行本次的回归预测任务
- 从0训练了神经网络并获得了接近state-of-art的成绩。
- 将项目部署到服务器。
- 数据源问题。对外公开的MRI图像资源非常有限,很多要去自己申请,并且下载下来的数据也需要进行预处理、清洗等操作。
- 选择图像处理工具问题。MRI图像的预处理复杂,本项目用到了5个步骤,每个步骤都有大量第三方工具来完成,如果选择的工具不恰当,将会对结果造成很大影响。
- 图像预处理的时间很长,平均一张图片大约需要6分钟时间左右完成预处理的过程。所以我当时直接用了10台服务器,将数据切成10块,让他们并行地跑,从而完成处理。
- CNN网络的选择问题。实际上我一开始没有想到直接用resnet来做,之前别人的做法,是VGG。我试了一下感觉效果不好,最后看到腾讯优图7月份发布的一篇医学图像分割的论文,才选择resnet来做这个事。(剪枝救命稻草)
- 训练模型的时间较长。因为训练模型需要指定一些像learning rate这样的超参,而这个超参需要不断地去炼丹、尝试才可以知道最优的参数。每一次尝试,可能都要花上一天时间来训练。好在南安普顿大学的GPU比较给力,不然项目铁定延期。
- 最后部署的困难。之前也说了,内存不够的问题,我不可能为了这个项目去把我的私人服务器升配到8G,毕竟又不是真的要投入使用。
- 思来想去,最终为什么没有达到state-of-art的成绩,大概跟数据集有关,state-of-art的论文在这儿:链接,论文里面用到的数据集有些我没采集到,所以我的training-set数量比他的要少。
- 在传统机器学习方法里,构建的特征比较简单,仅仅用了灰度直方图作为其特征,其实形状特征可能也很重要。
- 没有把脑灰质、脑白质、脑脊液分离开来。之前在state-of-art的论文里,作者提到,他是将三者分离开来喂到网络中,才有最终误差4.16 的成绩。而我是直接将整个脑部喂进去训练的。
- 没有尝试更多神经网络可视化的方法。NN有个通病就是解释性比较弱,导致它就是一个黑盒,出现了bad case也无从追溯其原因,而神经网络可视化可以瞥见其工作机制的一角,很遗憾我没有尝试更多的方法去做这方面的工作。除了我用的layer activations visualisation之外, 还有conv/fc filters visualisation, 和heatmaps of class activations可以去尝试。